はじめに
Rumaleにカーネル判別分析を追加して、これを version 0.18.4 としてリリースした。カーネル判別分析は、version 0.18.0 で追加したフィッシャー判別分析 (Fisher Discriminant Analysis, FDA) を、カーネル法により非線形化したものである。
rumale | RubyGems.org | your community gem host
使い方
Rumaleはgemコマンドでインストールできる。線形代数の計算にNumo::Linalg、実行結果の描画にNumo::Gnuplotを使いたいので、一緒にインストールする。
$ gem install rumale numo-linalg numo-gnuplot
Numo::Linalgのためにopenblasを、Numo::Gnuplotのためにgnuplotをインストールする。
$ brew install openblas gnuplot
カーネル判別分析は、教師あり次元削減手法とも捉えられる。一般に、カーネル判別分析により写像した部分空間で、k-近傍法で分類するとこが想定されている。Rumaleではfitメソッドで学習し、transformメソッドで次元削減を行う。
LIBSVM Dataで公開されているUCI Vowelデータセットを利用して、次元削減してk-近傍法で分類する例を示す。
カーネル判別分析では、カーネル関数の選択と正則化パラメータが、ハイパーパラメータとなる。カーネル関数自体にパラメータがあれば、それも調整する必要がある。
require 'numo/linalg/autoloader' require 'numo/gnuplot' require 'rumale' # 分類器には, 最近傍法を使用する. est = Rumale::NearestNeighbors::KNeighborsClassifier.new(n_neighbors: 1) # データセットを読み込み, 訓練・テストデータに分割する. x, y = Rumale::Dataset.load_libsvm_file('vowel.scale') x_train, x_test, y_train, y_test = Rumale::ModelSelection.train_test_split(x, y, test_size: 0.4, random_seed: 1) # 判別分析法で二次元空間に射影し, 部分空間で分類器の性能を評価する. fda = Rumale::MetricLearning::FisherDiscriminantAnalysis.new(n_components: 2) fda.fit(x_train, y_train) z_train = fda.transform(x_train) z_test = fda.transform(x_test) est.fit(z_train, y_train) fda_score = est.score(z_test, y_test) # カーネル判別分析法で二次元空間に写像し, 部分空間で分類器の性能を評価する. kfda = Rumale::KernelMachine::KernelFDA.new(n_components: 2, reg_param: 1e-8) gamma = 0.05 kmat_train = Rumale::PairwiseMetric.rbf_kernel(x_train, nil, gamma) kfda.fit(kmat_train, y_train) z_train = kfda.transform(kmat_train) kmat_test = Rumale::PairwiseMetric.rbf_kernel(x_test, x_train, gamma) z_test = kfda.transform(kmat_test) est.fit(z_train, y_train) kfda_score = est.score(z_test, y_test) # それぞれの正確度を出力する. puts "Accuracy (FDA): #{fda_score}" puts "Accuracy (KFDA): #{kfda_score}" # 訓練データセットの二次元空間を散布図としてプロットする. plots = y_train.to_a.uniq.sort.map do |l| [z_train[y_train.eq(l), 0], z_train[y_train.eq(l), 1], t: l.to_s] end Numo.gnuplot do set(terminal: 'png') #set(output: 'fda.png') set(output: 'kfda.png') plot(*plots) end
これを実行した結果が次のようになる。カーネル判別分析のほうが、優れた分類性能を得られている。
$ ruby kfda.rb Accuracy (FDA): 0.6445497630331753 Accuracy (KFDA): 0.9146919431279621
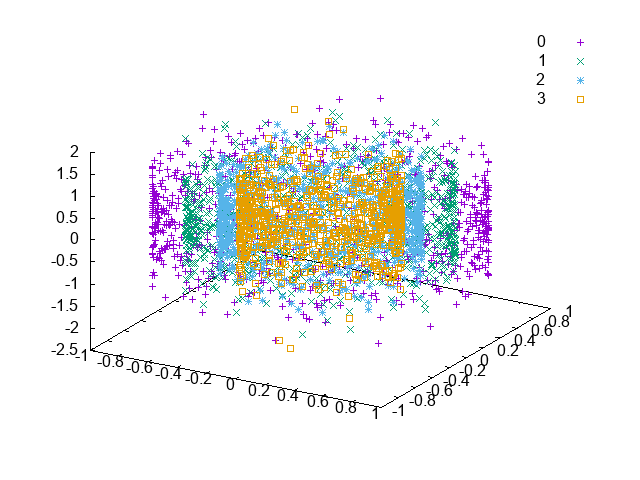
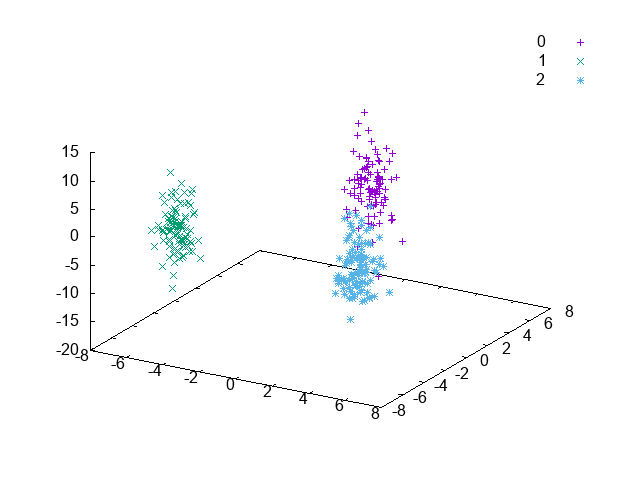
部分空間のサンプルを可視化したものが次のようになる。Vowelデータセットは、11クラスからなるデータセットだが、判別分析 (FDA) よりも、カーネル判別分析 (KFDA) のほうが、部分空間で同一クラスのサンプル同士が集まって位置していることがわかる。

どんなデータセットでも、このように上手く行くわけではないが、判別分析で高い分類性能が得られない場合などに、カーネル判別分析を試してみる価値はある。
おわりに
Rumaleに、version 0.18.0 で判別分析を追加したので、カーネル判別分析も追加してみた。カーネル判別分析は、非線形な教師あり次元削減手法でもあるので、多くの場合で判別分析よりも良い結果を得られると思う。一方で、カーネル関数の選択と、ハイパーパラメータの調整には気を配る必要がある。また、データセットが大きいと、カーネル関数の計算などで計算時間を必要とする。